实验室的研究侧重于结构生物学、基因组学、机器学习和大数据分析等交叉学科领域,主要研究方向是结合结构生物学和系统生物学,开发并使用计算与实验相结合的方法,解读生物大分子(如蛋白质、RNA★、DNA)的结构与功能关系,重建其相互作用网络★★★,发现与蛋白质和 RNA 结构变化,以及大分子相互作用异常相关的复杂疾病(包括癌症和传染性疾病)的发病机制和可能的治疗方法。
在「Meet AI4S」系列直播的第二期中,HyperAI超神经有幸邀请到了该研究成果的论文第一作者清华大学张强锋实验室博士后李雨哲,8 月 21 日,李雨哲博士将以线上直播的形式进一步为大家分享空间转录组学和单细胞组学研究中的 AI 方法。
* 开发了空间转录组数据人工智能分析工具 SPACE,可从单细胞分辨率的空间转录组数据中★★★,识别空间细胞类型并发现组织模块★★★。
为了进一步推进 AI4S 的普适化,将学术机构的科研成果进一步降低传播壁垒,分享给更广泛的行业学者、科技爱好者及产业单位★★,HyperAI超神经策划了「Meet AI4S」视频栏目,邀请深耕 AI for Science 领域的科研人员或相关单位★★,以视频的形式分享研究成果、方法思路,共同探讨 AI for Science 在科研进展及推进落地过程中面临的机遇和挑战,促进 AI for Science 的科学普及和传播★。
「Meet AI4S」系列直播第二期,邀请到了清华大学张强锋实验室博士后李雨哲。8 月 21 日,李雨哲博士将以线上直播的形式进一步为大家分享空间转录组学和单细胞组学研究中的 AI 方法★。
* SPACE 优于目前可用的工具,可用于从 ST 数据中区分空间信息细胞类型。
* SPACE 定义并发现了细胞社区,即一种其所组成的细胞类型的空间分布较为均质且具有可辨识边界的组织模块。
近年来,AI 浪潮涌向科研领域,也为空间转录组学与单细胞组学研究提供了创新思路。
* SPACE 在细胞类型识别和组织模块发现方面明显优于其他工具,尤其是在包含多种细胞类型的复杂组织中★★。
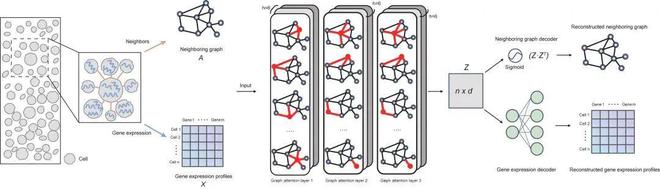
SPACE 使用图自编码器 (Graph autoencoder) 框架来学习低维的细胞嵌入,该细胞嵌入描述了空间转录组数据中每个细胞自身的基因表达信息以及其与空间邻近细胞的相互作用信息(因此称该细胞嵌入为细胞-细胞相互作用感知的细胞嵌入,cell-cell interaction-aware cell embedding)。在该细胞嵌入基础上,SPACE 再通过聚类算法识别空间细胞亚型和发现组织模块。
空间转录组技术是生物信息学领域近年来的重大突破之一,曾在 2020 年被 Nature Method 评为年度技术。
* SPACE 能够基于 ST 数据集中的空间信息识别出空间信息具有生物学差异的细胞类型。
本次直播将分享基因组学研究,主要是空间转录组学和单细胞组学研究中的人工智能方法。
基于空间转录组技术★★,既能获得高分辨率的转录组数据,还能和位置信息相对应★,确定不同细胞亚型或转录状态在空间上的分布和位置关系。
随着空间转录组技术的不断发展与迭代★★,研究者能够在单细胞分辨率下获得细胞的基因表达谱,同时保留细胞在组织内的空间位置信息。如何有效地利用这些空间信息来识别空间细胞亚型并发现组织模块,成为空间转录组数据分析的核心任务★。
3. 了解基于图神经网络的空间转录组学数据表征算法 SPACE 和细胞社区概念。
从架构来看,SPACE 模型由三部分组成★★:编码器 (三层图注意网络)、邻近图解码器和基因表达解码器,下图显示了该模型的整体框架★★★:
张强锋实验室隶属于清华大学生命科学学院,同时也是清华-北大生命科学联合中心★★★、北京结构生物学高精尖创新中心的重要组成部分。
* 细胞社区是由其所组成的细胞间相似的相互作用网络定义的,这种相互作用网络可以用于优化基于配体-受体的细胞通讯推断。
随着生命科学研究和人工智能 (AI) 技术的快速发展,AI 在生物医学研究中正发挥越来越重要的推动作用,产生了「AI for Science」的交叉研究新范式★★★。
实验室拥有独特的蛋白质和 RNA 结构建模、基于新一代测序的 RNA 结构测量★★、高通量 RNA 蛋白相互作用检测技术,以及强大的计算和实验平台,以推进研究人员的前沿研究。
例如★★,清华大学生命科学学院张强锋副教授课题组,开发了基于图自编码器深度学习框架的人工智能算法 SPACE,能够从单细胞分辨率的空间转录组数据中识别空间细胞类型和发现组织模块★,可被用于大规模的空间转录组研究。
* SPACE 可用于大规模的空间转录组研究,以了解空间邻近细胞之间的相互作用如何影响细胞类型和组织模块生物学功能★★。
